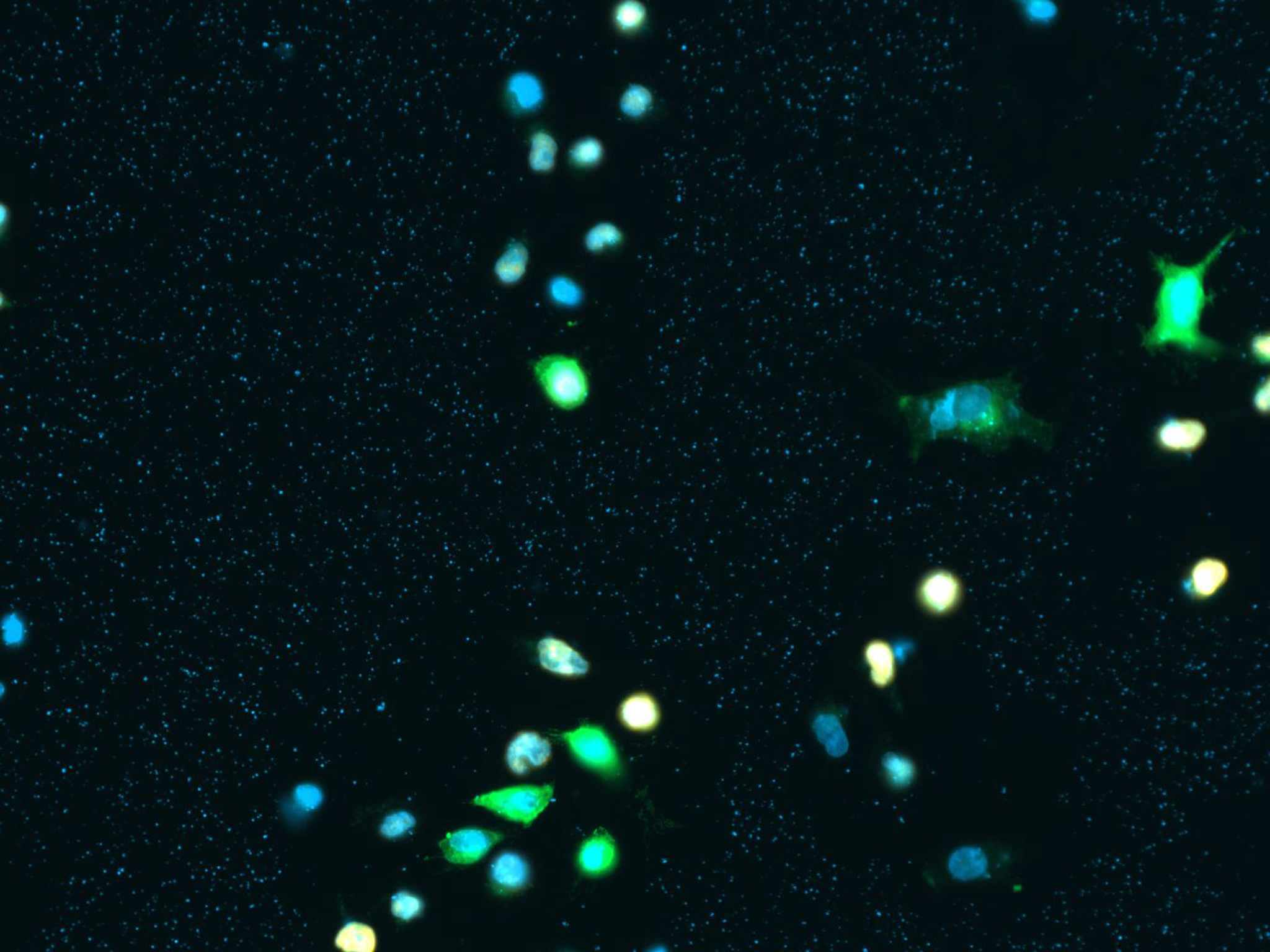
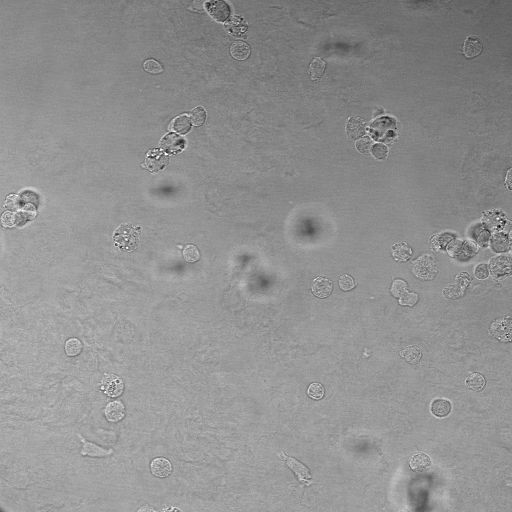
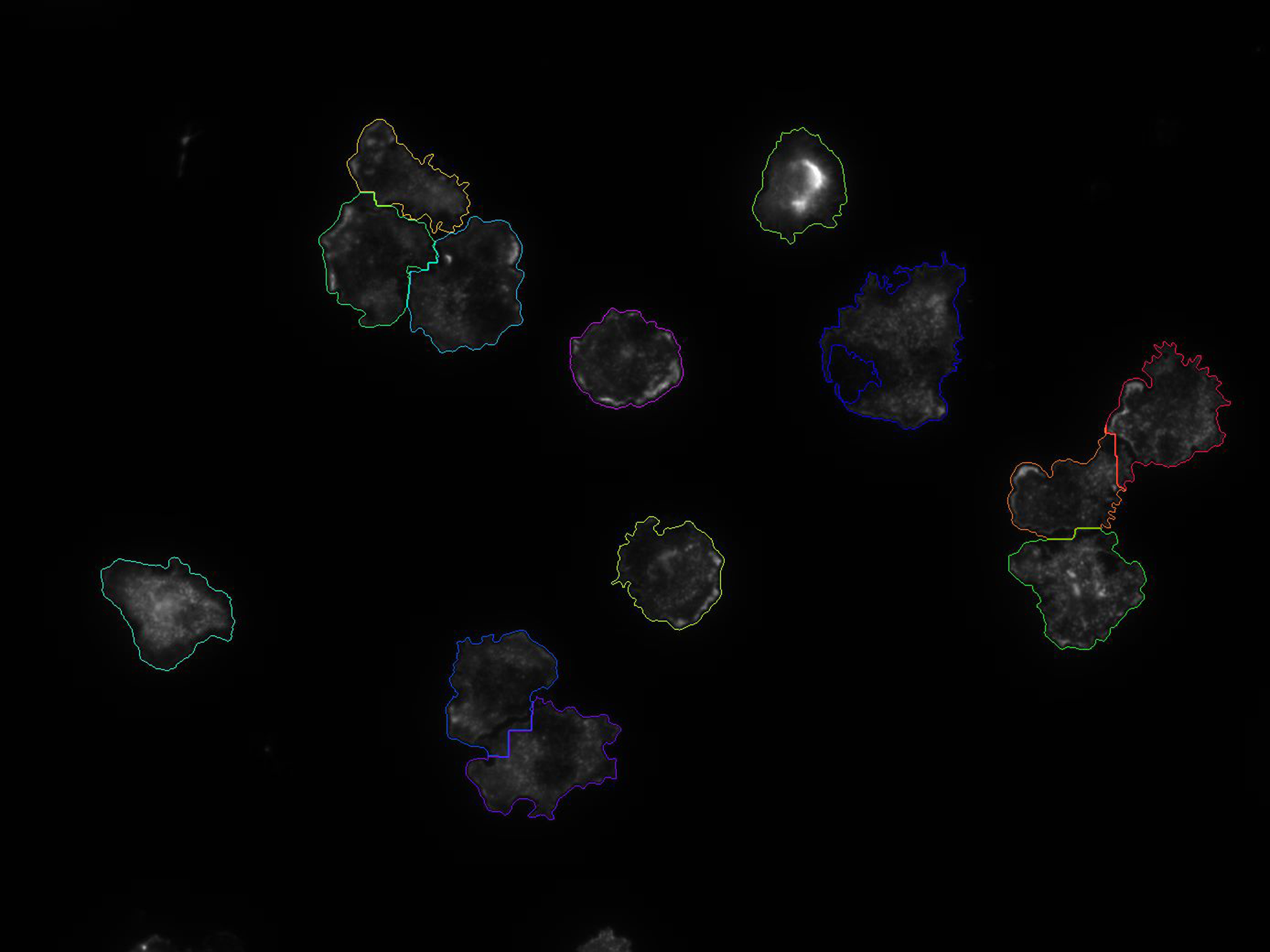
Proliferation und Kolokalisation
Problem:
Das Zählen von Zellen, die verschiedene fluoreszent markierte Proteine exprimieren, gehört zum Alltag der meisten Forscher im Bereich der Biowissenschaften. Die manuelle Auswertung ist allerdings langwierig, fehleranfällig und wegen der inter- und intraindividuellen Untersuchungsvariabilität nicht immer reproduzierbar.
Unsere Lösung:
Automatische Analyse der Fluoreszenz-Bilder und Ausgabe eines Excel-Datensatzes
Aktueller Status:
- Erfolgreich in der wissenschaftlichen Praxis eingesetzt:
Unterer B, et al. IFN--response mediator GBP-1 represses human cell proliferation by inhibiting the Hippo signaling transcription factor TEAD. Biochem J. 2018 Aug 17 - Eigenständige Software als Prototyp verfügbar
- C/C++ API in Entwicklung
Unsere Lösung:
- Umfangreiche modulare Toolbox mit ca. 50 Bildanalyse-Bausteinen verfügbar. Wir kombinieren und justieren diese Bausteine, um leistungsfähige Bildanalyse-Algorithmen zu erstellen, die als Vorlage/Presets gespeichert werden können
- Unsere Lösung ist KI-frei und benötigt daher keine große Datenbank mit annotierten Grundwahrheiten
- Optional kann die Bildanalyse-Pipeline auch automatisch optimiert werden, indem man die Grundwahrheit durch wenige annotierte Bilder vorgibt