Unsere Motivation – Effizienz und Reproduzierbarkeit
Forschung und Entwicklung in den »Life Sciences« wie in der Pharmakologie, der Mikrobiologie, der Virologie und der Immunologie erfordern eine fluoreszenzbasierte Auswertung von Zellen unter dem Mikroskop. Dabei kommen oftmals mehrere Fluoreszenzkanäle zum Einsatz, die verschiedenste Zellstrukturen (Zellplasma, Zellkerne etc.) abbilden.
Die Auswertung und Interpretation solcher meist sehr umfangreichen Datensätze ist nach wie vor ein zeit- und arbeitsaufwändiger manueller Prozess. Mangelnde Reproduzierbarkeit durch subjektive und tagesformabhängige Bewertung können die Interpretation beeinflussen und die Aussagekraft von Ergebnissen beeinträchtigen.
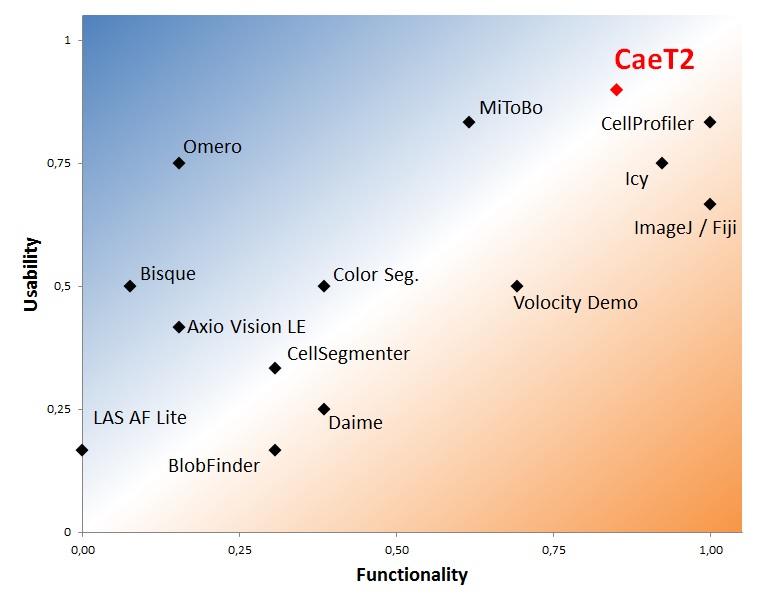
Hohe Datenqualität und Reproduzierbarkeit wie auch eine effiziente und wirtschaftliche Analyse werden auch in der Forschung über den Erfolg entscheiden. An dieser Stelle unterstützt das Fraunhofer-Institut für Integrierte Schaltungen IIS durch leistungsfähige und maßgeschneiderte Software-Lösungen für eine rechnergestützte Fluoreszenzbildauswertung.